| Doctor Stat |
Генетический анализ предрасположенности к заболеваниям
- Выявление аллелей
- Выявление генотипов для одного гена
- Выявление генотипов для нескольких генов
- Отклонение частот генотипов от равновесия Харди-Вайнберга
1.Выявление аллелей
Мы ищем сочетания аллелей одного гена, ведущих к болезни непосредственно или через предрасположенность. В учебной базе данных заведено 3 гена, см.рис.1
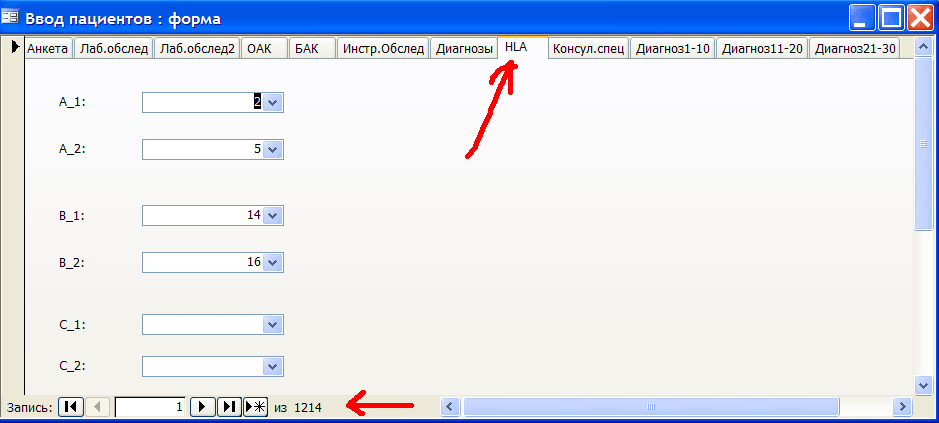
Рис.1 Ввод генотипов для пациента в базе данных.
Первый ген (слева вверху) называется A. Потом идет подчеркивание и номер аллеля. Для каждого пациента вводится 2 аллеля: A_A1 и A_A2 по числу гомологичных хромосом. На рис.1 пациент гетерозиготен по гену А и имеет аллели 2 и 5. Придерживайтесь правила ввода гетерозигот: номер 1-ого аллеля не должен быть больше номера 2-ого. Генотипирование по гену С для данного пациента не проводилось - поля остались незаполненными. На рис.1 показан 1-ый пациент из 1214.
1.1 Перебор аллелей вручную
Мы хотим определить, отличается ли частота одного или группы аллелей между выборками больных и здоровых? Выберем какую-либо болезнь. Пусть это будет стенокардия. Заметим, что стенокардия делится на 2 вида: Стенокардия напряжения и Спонтанная (вариантная, вазоспастическая) стенокардия. В нашу выборку попадут оба вида. В качестве сравниваемых групп сконструируем 2 выборки пациентов в возрасте от 40 до 50 лет и протестируем их на наличие в базе:
- стенокардия есть в диагнозах (25 чел.)
- стенокардии нет (248 чел.)
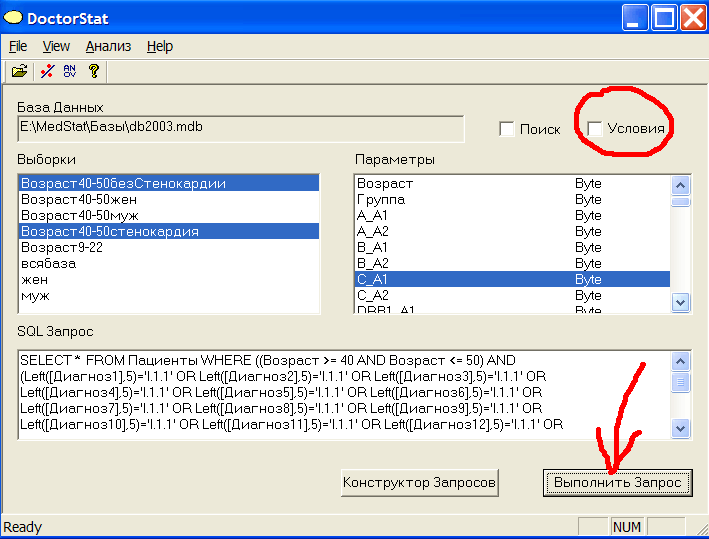
Рис.2 Сравнение аллелей гена C спомощью хи-квадрат.
В Excel появится два листа: с гистограммой и с результатами теста хи-квадрат. На гистограмме показаны частоты аллелей гена C в 2-х выборках, см.рис.3
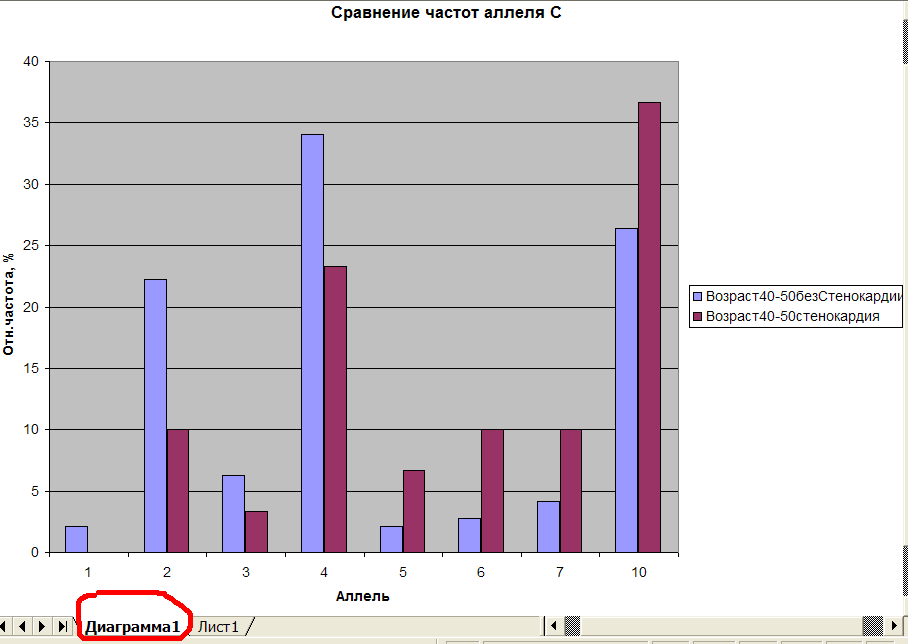
Рис.3 гистограмма частот аллелей гена C.
Из рис.3 видно, что 1-ой аллель в группе больных стенокардией не встречается и его частота меньше 5%, т.е. он редкий аллель. В группе контроля наиболее часто встречается аллель 4. В группе больных наиболее часто встречается аллель 10. Посмотрим результаты теста хи-квадрат на Лист1, см.рис.4
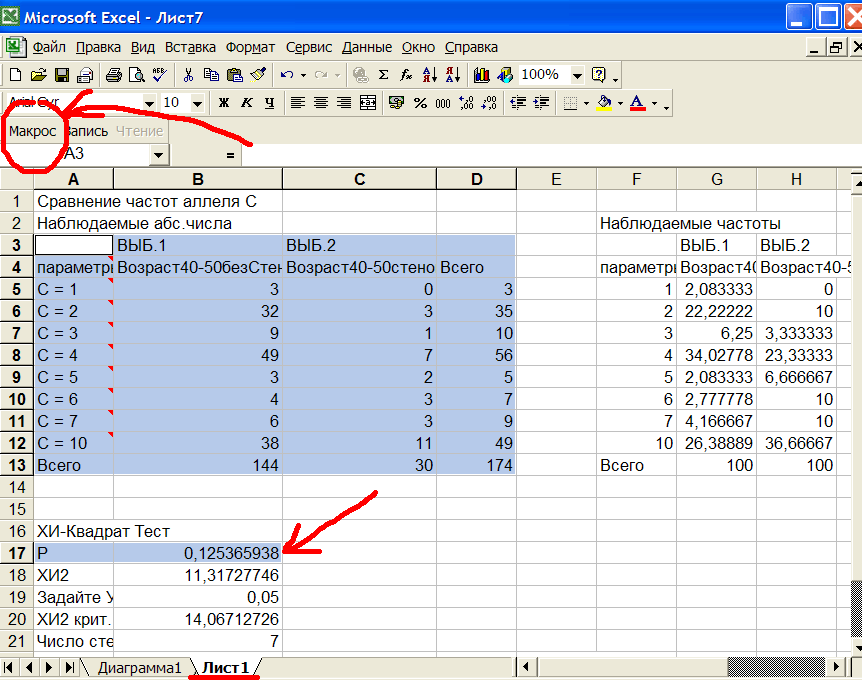
Рис.4 Результат хи-квадрат теста всех аллелей гена C.
На рис.4 в первом столбце (А) выведены номера аллелей. Во 2-ом столбце (B) выведены количества аллелей в выборке здоровых. В 3-м столбце (С) выведены количества аллелей в выборке больных стенокардией. Из рис.4 видно, что хотя частоты отдельных аллелей отличаются больше чем в 2 раза, а некоторые аллели (С=1) встречаются только в первой выборке ВЫБ.1, значимость теста хи-квадрат равна P=0,125(ячейка B17), что больше критического уровня значимости 0,05. Отсюда можно сделать вывод: тест хи-квадрат не выявил различий в частотах отдельных аллелей между 2-мя выборками. Вы можете возразить, что некоторые ожидаемые числа в таблице слишком малы (<1), см.рис.5, поэтому тест хи-квадрат применять нельзя.
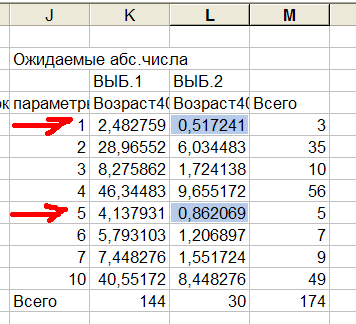
Рис.5 Таблица ожидаемых чисел аллелей гена C.
Из рис.5 видно, что ожидаемые числа для аллелей 1 и 5 ВЫБ.2 меньше 1. Существует два способа решения этой проблемы. Первый способ - объединять аллели в группы, тем самым увеличивая ожидаемые числа. Второй - исключить редкие аллели из анализа. Применим 2-ой способ и исключим аллели 1,5. Для этого выделим выборки и один из аллелей гена С и выберем в меню Гены->Хи-квадрат->Ручной или нажмем клавишу Ctrl+U, см.рис.5а
Рис.5а Ручной хи-квадрат аллелей гена C.
Появится новое окно, см.рис.6:
Рис.6 Ручной выбор аллелей гена C.
Выделим в левом окне с помощью Ctrl все аллели, кроме 1,5 и нажмем кнопку ХИ КВАДРАТ. В Excel появится гистограмма без 1 и 5 аллелей, и на Лист1 выведутся результаты теста хи-квадрат, см.рис.7
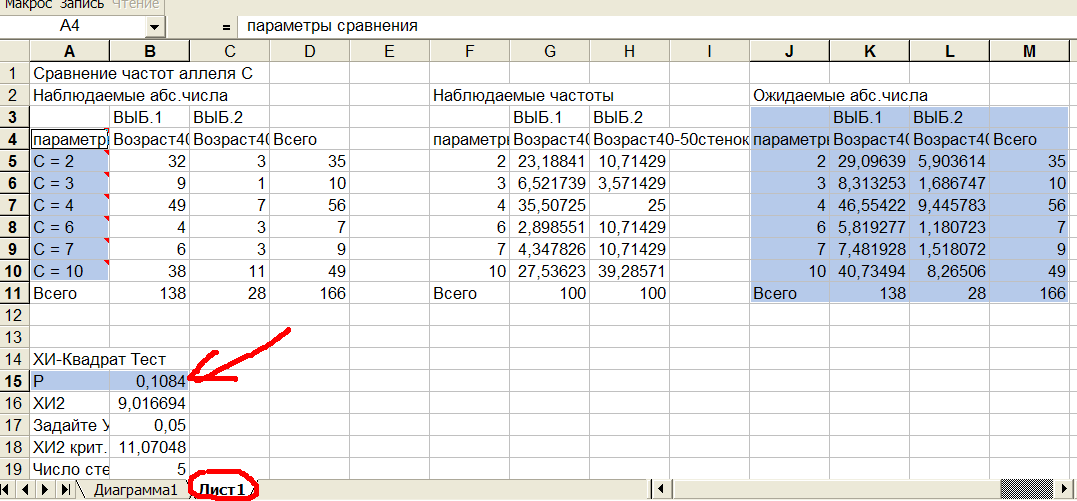
Рис.7 Результаты сравнения выборочных аллелей гена C.
Из рис.7 видно, аллели 1 и 5 исчезли из столбца А. Все ожидаемые абсолютные числа (таблица справа) больше 1, поэтому тест хи-квадрат применим. Р-value (ячейка B15) уменьшилось с 0,125(см.рис.4) до 0,1084 > 0,05. Результат сравнения остался прежним: выборки не отличаются.
Исключая редкие аллели, мы получили некоторое увеличение значимости отличий. Попробуем теперь объединять аллели с близким отношением частот, увеличивая абсолютные числа и, тем самым, повышая значимость различий выборок.
1.2 Автоматическая кластеризация аллелей
Попробуем применить кластеризацию, чтобы найти различия между выборками здоровых (контроль) и больных по частотам аллеля гена С. Кластеризация объединяет аллели с приблизительно одинаковым отношением частот. Посмотрите на гистограмму рис.3. Аллели 1, 2, 3, 4 чаще встречаются в выборке здоровых, а 5, 6, 7, 10 - в выборке больных. Разумно объединить первую группу аллелей в один кластер (здоровых аллелей), 2-ую группу - в другой кластер (больных аллелей). С помощью критерия хи-квадрат показать, что каждый кластер однороден по частотам аллелей, а между кластерами существует значимое отличие. Весь этот анализ проводится автоматически при нажатии кнопки Макрос, см.рис.4 или комбинации клавиш Ctrl+M(англ.) в Excel. Результаты кластеризации приведены на рис.8
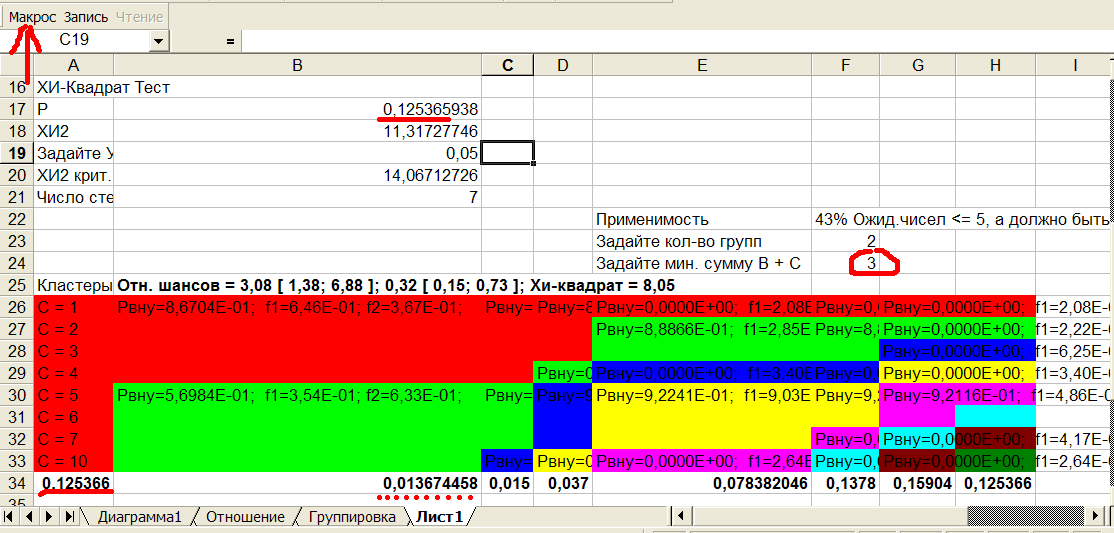
Рис.8 Результат кластеризации аллелей гена C.
При нажатии кнопки Макрос под таблицей результатов теста хи-квадрат выводится результат кластеризации, т.е. объединения аллелей в группы, выделенные одним цветом. Столбец А, в котором показаны номера аллелей, выделен красным цветом - это один кластер. Столбец В выделен 2-мя цветами: красным и зеленым (два кластера). Столбец С - 3 кластера и т.д. В последнем столбце G каждый аллель покрашен в свой цвет, т.е. является отдельным кластером. Ячейка F24 (Задайте мин. сумму B + C) является ячейкой ввода. Мы изменили минимальную сумму с 5 (по умолчанию) на 3, чтобы редкий 1-ый аллель попал в таблицу кластеризации. Наоборот, увеличивая минимальную сумму, мы выбрасываем редковстречающиеся аллели из анализа. Попробуйте ввести 5 и нажать кнопку Макрос, чтобы исключить 1-ый аллель из кластерного анализа.
Как мы и ожидали, алгоритм собрал здоровые аллели 1,2,3,4 в один кластер (красный), а больные аллели 5,6,7,10 - в другой (зеленый), см.2-ой столбец B. Но что значат столбцы С,D,..., в которых число кластеров равно 3,4,...? Эти подкластеры свидетельствуют о неоднородности выборок и описывают различные подвиды здоровья и болезни.
Внутри каждого кластера выводится три числа: внутригрупповая вероятность, частота в 1-ой выборке, частота во 2-ой выборке. Например, ячейка B26 внутри красного кластера. Внутригрупповая вероятность Рвну=0,867, что много больше 0,05. Это говорит о высокой однородности кластера. Частота набора красных аллелей 1,2,3,4 в первой выборке f1=0,646 больше, чем частота во второй выборке f2=0,367. Следовательно, красный набор аллелей способствует здоровью, т.к. 1-ая выборка контрольная (без стенокардии). Зеленый набор (кластер) аллелей 5,6,7,10 менее однороден Рвну=0,570 (ячейка B30), соответственно f1=0,354 < f2=0,633, т.е. зеленый набор аллелей чаще встречается во 2-ой выборке (больных), поэтому мы говорим, что зеленые аллели способствуют стенокардии. Сумма каждой частоты f1 и f2 для любого столбца =1. Проверим для f1(столбец B): 0,646+0,354=1.
Под каждым столбцом выводится межгрупповая (межкластерная) вероятности с учетом множественных сравнений. Например, возьмем столбец B таблицы кластерного анализа. Под ним, в ячейке B34 выведена p-value=0,0137 < 0,05 сравнения 2-х наборов аллелей (красных и зеленых). Мы получили два кластера, по которым выборки значимо различаются. А что можно сказать о большем количестве кластеров? С увеличением количества кластеров от 2 до 7 (столбцы B-G) p-value увеличивается, поэтому значимость межкластерных различий падает:
- 3 кластера p-value=0,015(ячейка С34) < 0,05 - еще отличаются
- 4 кластера p-value=0,037(ячейка D34) < 0,05 - отличаются, но меньше
- 5 кластеров p-value=0,078(ячейка E34) > 0,05 - уже НЕ отличаются
- красного кластера: шанс встретить красные аллели в 1-ой выборке в 3,08 раза больше, чем во 2-ой выборке
- зеленого кластера: шанс встретить зеленые аллели во 2-ой выборке в 3,08 раза больше, чем в 1-ой выборке
- красного кластера: шанс встретить красные аллели во 2-ой выборке в 0,32=1/3,08 раза больше, чем во 1-ой выборке
- зеленого кластера: шанс встретить зеленые аллели в 1-ой выборке в 0,32=1/3,08 раза больше, чем в 2-ой выборке
Доверительный интервал в квадратных скобках: 3,08 [ 1,38; 6,88 ] показывает каким может оказаться отношение шансов в популяции с вероятностью 95%. Мы видим, что единица не входит в доверительный интервал, значит найденное различие статистически значимо.
2.Выявление генотипов для одного гена
Мы ищем сочетания генотипов одного гена, ведущих к болезни непосредственно или через предрасположенность.
2.1 Перебор генотипов вручную
Мы хотим определить, отличается ли частота одного или группы генотипов между выборками больных и здоровых? Выберем ту же самую болезнь, что мы использовали при анализе аллелей - стенокардию и те же 2 группы (выборки) пациентов. Если вы еще их не создали, то сконструируем 2 выборки пациентов в возрасте от 40 до 50 лет:
- стенокардия есть в диагнозах (25 чел.)
- стенокардии нет (248 чел.)
Рис.9 Сравнение генотипов гена C.
В Excel появится два листа: с гистограммой и с результатами теста хи-квадрат. На гистограмме показаны частоты генотипов гена C в 2-х выборках: синяя - контроль, красная - больные, см.рис.10
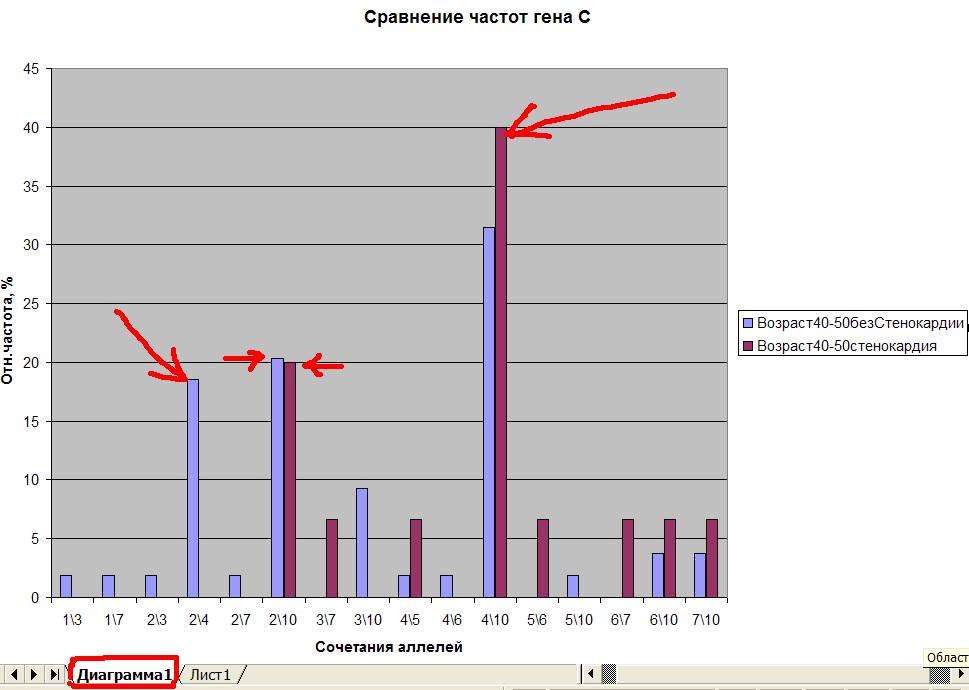
Рис.10 Гистограмма частот всех генотипов гена C.
Сравнивая рис.10 и рис.3, видно, что количество генотипов гена С в 2 раза больше чем количество аллелей. Наиболее часто встречающийся генотип в группе здоровых(контроля) и больных один и тот же 4\10. Генотип 2\4 имеет высокую частоту 18% в группе контроля и не встречается в группе больных. Посмотрим результаты теста хи-квадрат на Лист1, см.рис.11
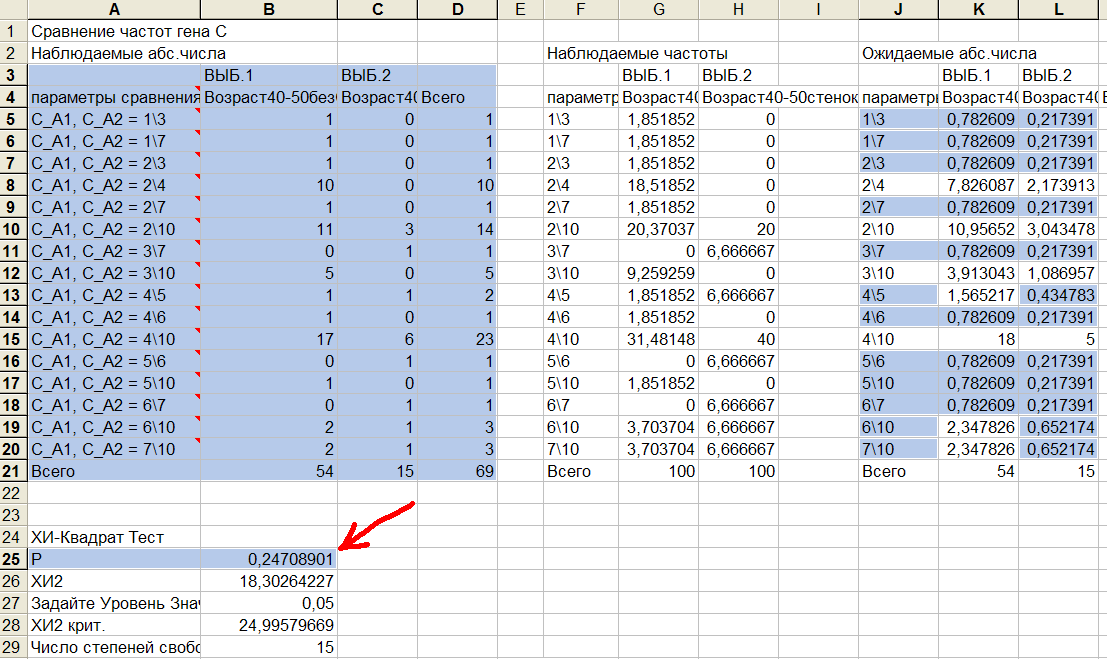
Рис.11 Результат хи-квадрат теста всех генотипов гена C.
На рис.11 в первом столбце (А) выведены названия генотипов. Во 2-ом столбце (B) выведены количества пациентов в контрольной выборке Возраст40-50безСтенокардии. В 3-м столбце (С) выведены количества пациентов в выборке больных стенокардией. Внизу (строка 25) выведена p-value=0,247>0,05. Вывод: хи-квадрат тест не выявил различий в частотах генотипов. В таблице Ожидаемые абс.числа (справа) из-за наличия редких генотипов присутствует очень много малых значений (<1), поэтому применимость хи-квадрат теста вызывает сомнения.
Исключим из анализа редкие генотипы. Для этого выделим выборки и ДВА аллеля гена С (см.рис.9), но нажмем не кнопку Выполнить Запрос, а выберем в меню Гены->Хи-квадрат->Ручной, (см.рис.5а). Появится новое окно, см.рис.12
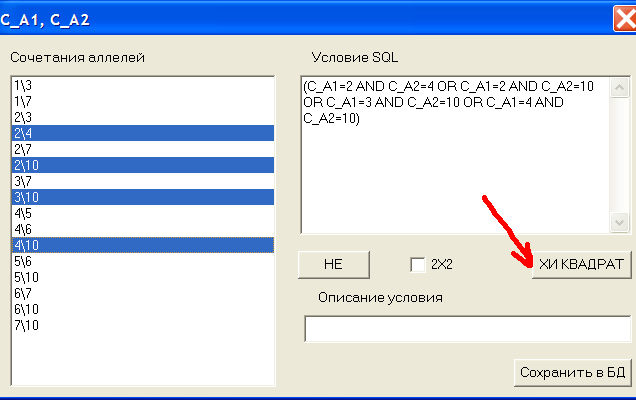
Рис.12 Ручной выбор генотипов гена C.
Выделим в левом окне с помощью Ctrl все генотипы, ожидаемые числа которых >1 (см.рис.11) 2\4,2\10,3\10,4\10 и нажмем кнопку ХИ КВАДРАТ. В Excel появится гистограмма, см.рис.13
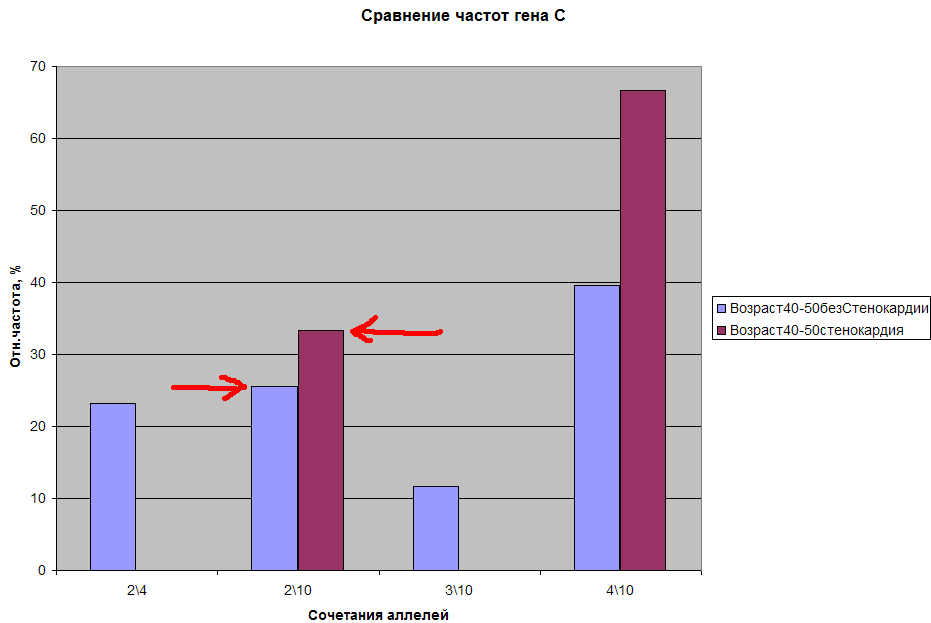
Рис.13 Гистограмма генотипов с ожидаемыми числами >1.
Сравнивая рис.13 и рис.10, видим, что на гистограмме всех аллелей (рис.10) частота генотипа 2\10 больше для выборки контроль, а на гистограмме частовстречающихся аллелей (рис.13) наоборот. Эффект изменения отношения частот при добавлении или исключении генотипов объясняется различным изменением частот для разных выборок. Посмотрим на результат теста, рис.14
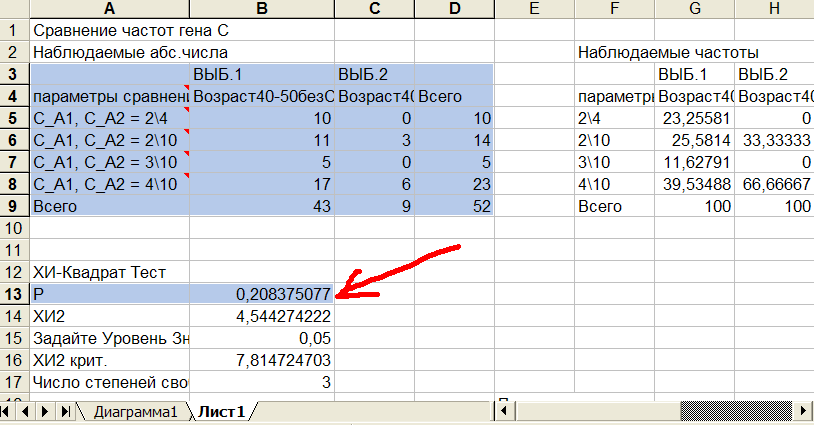
Рис.14 Результаты сравнения наиболее часто встречающихся генотипов гена C.
Сравнивая рис.14 и рис.11 видно, что значимость отличий после исключения редких генотипов возросла (P уменьшилось с 0,247 до 0,208), но все еще недостаточна для критического уровня: Р=0,208(ячейка B13)>0,05. Попробуем объединять близкие генотипы, как мы это делали с аллелями, т.е. применим кластеризацию.
2.2 Автоматическая кластеризация генотипов
В разделе 2.1, рассматривая каждый генотип отдельно, мы вычислили значимость различий между выборками контроля и больными. С помощью исключения редких генотипов значимость отличий была увеличена. Цель кластеризации - найти группы генотипов (аллелей, условий), как можно более однородные внутри себя и как можно более отличные между собой. Выделим выборки и генотипы, как на рис.9 и нажмем кнопку Выполнить Запрос. В Excel появится гистограмма частот генотипов, см.рис.10. Нажимаем кнопку Макрос для выполнения кластеризации и получаем рис.15
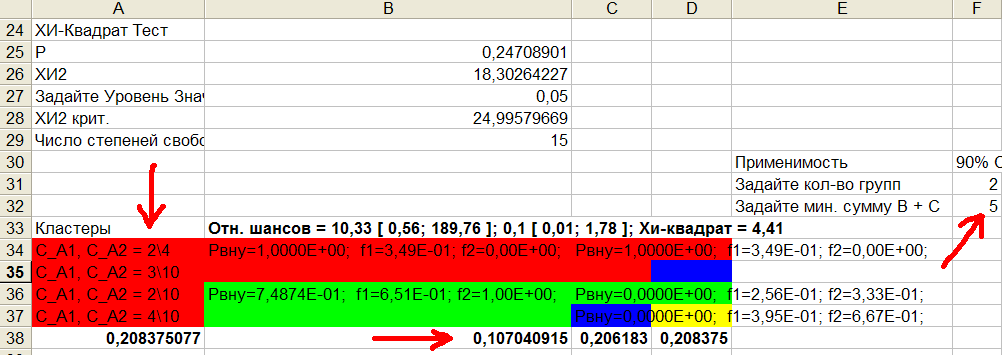
Рис.15 Результаты кластеризации генотипов гена C.
Сравнивая рис.14 и рис.15, видим, что алгоритм кластеризации автоматически оставил генотипы: 2\4, 3\10, 2\10, 4\10, которые раньше нам пришлось выбирать вручную по таблице ожидаемых чисел. Величина P уменьшилась почти в 2 раза с 0,208 (рис.14) до 0,107 (ячейка B38, рис.15). Таким образом, объединяя отдельные генотипы в группы мы сильно уменьшаем P, тем самым сильно повышая значимость отличий. Несмотря на заметное уменьшение Р, нам не удалось преодолеть критический порог значимости 0,05, как это случилось при анализе аллелей. Вывод: на 5%-ом уровне значимости группы контроля и больных не отличаются по генотипам гена С.
3.Выявление генотипов для нескольких генов
В предыдущем разделе мы пытались найти сочетания генотипов одного гена, повышающих риск заболевания (стенокардии). Сейчас нас будет интересовать влияние нескольких генов на болезнь.
3.1 Вычисление доли гена в заболевании
Предположим, что нам нужно организовать лабораторию диагностики предрасположенности к какой-нибудь болезни (например, стенокардии) и найти подходящие гены-кандидаты. В учебной базе данных хранятся 3 гена: А, В и С, см.рис.1. Мы хотим знать, есть ли связь между нашими генами и болезнью? Если связь существует, то на какие критерии следует обращать внимание при выборе гена? Один из важных параметров - значимость разделения кластеров генотипов (межгрупповая вероятность), которую мы рассмотрели выше. Но одной значимости недостаточно, т.к. высокозначимый генотип может очень редко встречаться в популяции. Второй параметр, на который следует ориентироваться - доля гена в болезни. Гены, вносящие малый вклад в заболевание, не подходят для диагностики, т.к. описывают малую часть больных. Генотипы гена С, как было показано, не влияют на стенокардию, поэтому долю этого гена в стенокардии вычислять не будем. Займемся 2-мя оставшимися генами А и В.
Сначала проведем автоматическую кластеризацию генотипов гена А, см.рис.16
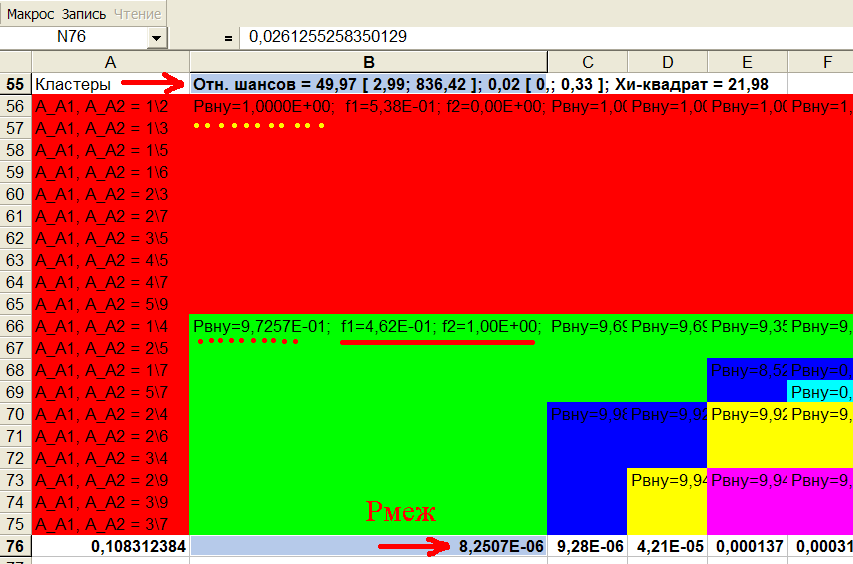
Рис.16 Результаты кластеризации генотипов А.
Из рис.16 видно, что полученные два кластера генотипов - красный и зеленый внутри высокооднородны: Рвну=1,00 (В56) для красного кластера и Рвну=0,97 (В66) для зеленого кластера. Межгрупповая вероятность (ячейка В76) Рмеж=8,2507Е-6 < 0,05, что говорит о высокозначимом различии между кластерами. Отношение шансов встретить зеленые генотипы во 2-ой выборке (больных) по отношению к 1-ой выборке (контроль) равно 49,97>1 (ячейка В55). В доверительный интервал для отношения шансов единица не входит: [2,99; 836,42], что еще раз подтверждает значимость различий 2-х кластеров.
Красный кластер генотипов - это здоровый кластер, т.к. его частота в контрольной выборке больше, чем в больной: f1=0,538>f2=0 (не встречается в больной выборке). Сохраним зеленый кластер генотипов гена А, см.рис.16, чаще встречающихся в группе больных (f2=1>f1=0,462), в базе данных. Для этого щелкнем мышью внутри любой ячейки зеленого кластера столбца В, например в ячейке В66, и нажмем кнопку Запись (справа от кнопки Макрос). В Excel2007 нужно нажать Ctrl+Shift+M (англ.раскладка клавиатуры). Появится окно ввода, в котором нужно ввести с клавиатуры название условия. Я ввел стенокардия_А. Нажать кнопку ОК, после чего это условие попадет в базу данных, см.рис.17:
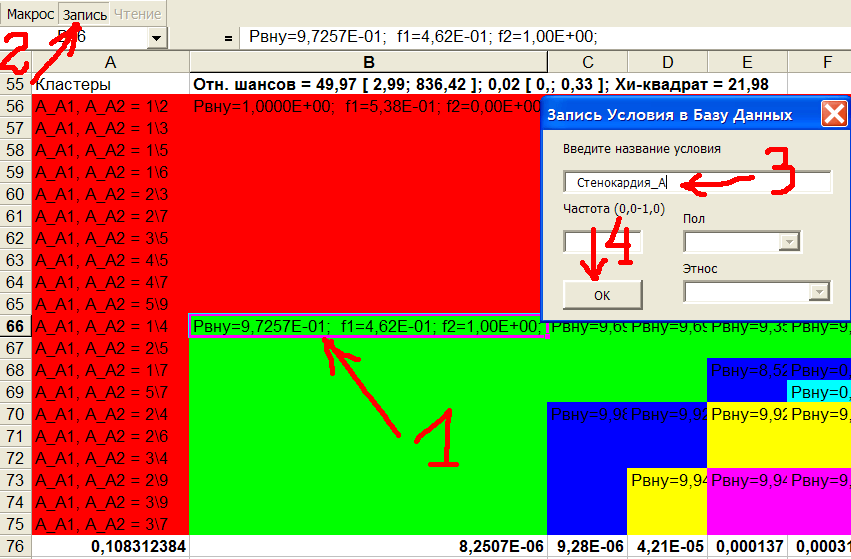
Рис.17 Запись больного кластера гена А в базу данных.
Проделаем аналогичную кластеризацию и сохраним кластеры больных генотипов для гена В, см.рис.18:
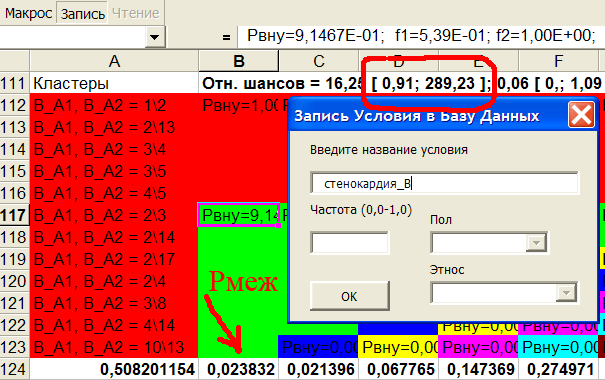
Рис.18 Запись больного кластера гена В в базу данных.
Из рис.18 видно, что межгрупповая вероятность (ячейка В124) Рмеж=0,023832 < 0,05, что говорит о значимом отличии 2-х кластеров, хотя значимость гена В меньше значимости А. С другой стороны, 1 попадает в доверительный интервал для отношения шансов (рис.18, строка 111), что говорит об отсутствии различий. Полученное противоречие вызвано разным способом расчета вероятностей. Оставим ген В в анализе, т.к. межгрупповая вероятность значима.
Чтобы вычислить доли генов А и В в заболевании стенокардией, выделим (с клавишей Ctrl) СНАЧАЛА контрольную выборку Возраст40-50безСтенокардии, ПОТОМ больную выборку Возраст40-50стенокардия, поставим галку в окне Условия и выделим 2 условия: стенокардия_А и стенокардия_В, отвечающие за кластеры больных генотипов соответствующих генов, нажмем кнопку Регрессия, см.рис.19
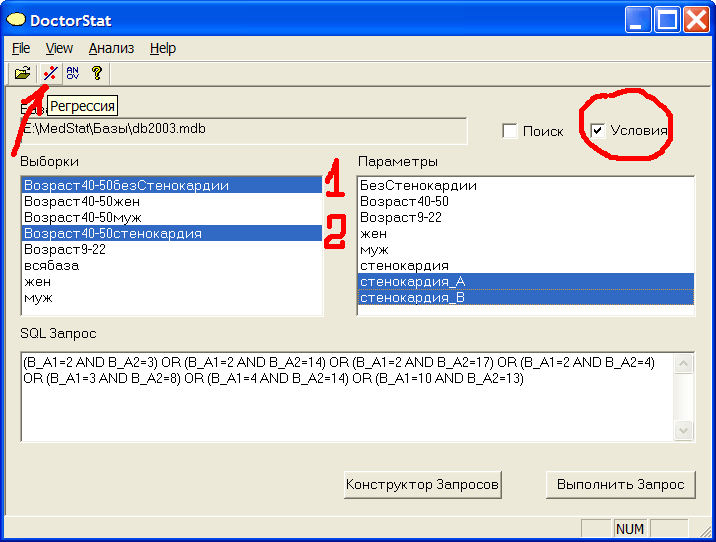
Рис.19 Подготовка вычислений доли генов в стенокардии.
В Excel появится таблица из 0 и 1. Каждая строка соответствует отдельному пациенту. В первом столбце выводится Признак болезни: 1 - болезнь есть, 0 - нет. В 2-ом и следующих столбцах с названиями условий стенокардия_А и стенокардия_В выводится 1, если условие выполнено и 0, если нет. Например, в строке 10 находится: 1 1 0. Это значит, что пациент больной (имеет в диагнозе стенокардию), его генотип входит в больной кластер гена А и здоровый кластер гена В. Нажмите кнопку Макрос и увидите результаты вычислений, см.рис.20:
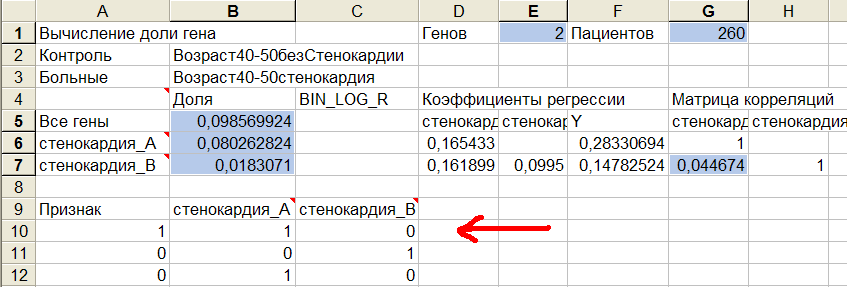
Рис.20 Результаты вычислений доли генов А и В в стенокардии.
Из рис.20 видно, что количество анализируемых генов=2 (Е1), количество пациентов, генотипированных по генам А и В =260 (G1). Доля больных пациентов, которую можно объяснить всеми генами, участвующими в анализе=0,0985 (В5). Другими словами, стенокардия на 9,85% определяется генами А и В. Эта доля складывается из вкладов отдельных генов. Гены располагаются по высоте таблицы в зависимости от вклада: чем выше вклад, тем выше располагается ген. Например, ген А с вкладом 0,080, располагается над геном В с вкладом 0,018. Значимость и вклад в болезнь для гена А больше, чем для гена В, поэтому ген А важнее для диагностики, чем ген В. Справа показана матрица корреляций, из которой следует, что кластеры генов А и В слабо коррелированы, т.к. коэффициент их корреляции=0,044674, что много меньше 1, значит гены независимы, и их отдельные вклады можно суммировать.
3.2 Вычисление риска заболевания
В п.3.1 мы проранжировали гены по важности для диагностики предрасположенности к стенокардии. Но как вычислить риск заболевания для конкретного пациента? Для этого нужно использовать логистическую регрессию, алгоритм которой реализован во многих статистических программах, например в SPSS. Копируем таблицу нулей и единиц из Excel, см.рис.20 в SPSS, выбираем Analyze->Regression->Binary Logistic…. В качестве Dependent выбираем var00001. В качестве Covariates выбираем var00002,var00003. Выбираем Method Backward LR. Нажимаем кнопку ОК, см.рис.21:
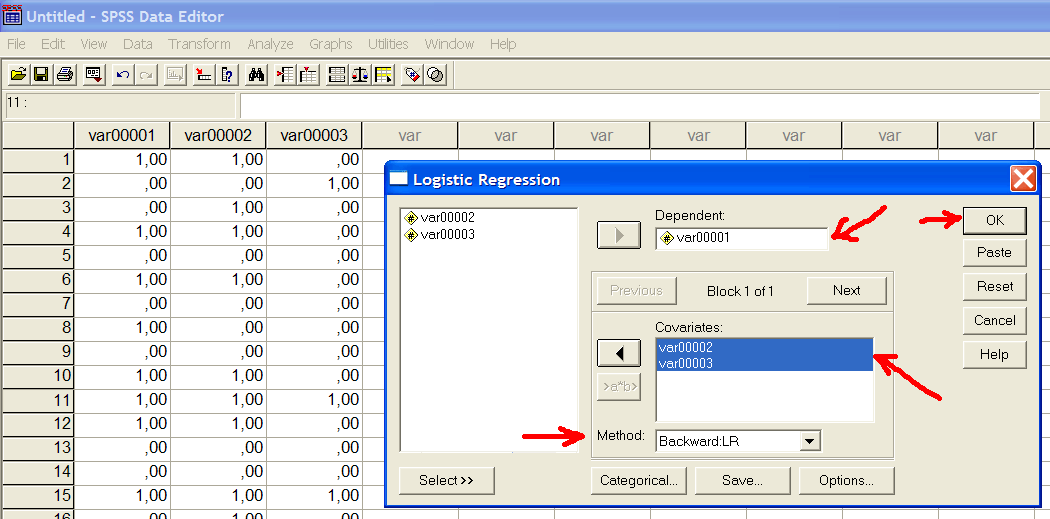
Рис.21 Подготовка к логистической регрессии в программе SPSS.
Результат логистической регрессии показан на рис.22:
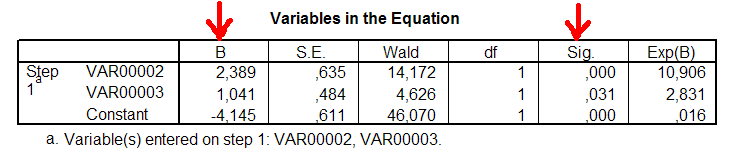
Рис.22 Результат логистической регрессии.
Напомню, что var00002 - переменная гена А, var00003 - переменная гена В. Обе переменные значимы, т.к. Sig(А)=0,000< 0,05, Sig(B)=0,031< 0,05, что согласуется с кластерным анализом, см.рис.16, рис.18. Уравнение регрессии будет иметь вид:
z=−4,145+2,389×A+1,041×B
, где переменные А и В принимают значения 0 (здоровый кластер) или 1 (больной кластер) для генов А и В соответственно.Для вычисления риска заболевания используется формула:
P=(1+exp(−z))−1
3.3 Выявление взаимодействия генов
В п.3.1 показано, что гены А и В по отдельности значимо влияют на стенокардию. Корреляция между кластерами этих генов мала, см.рис.20. Существует ли взаимодействие между генами? Изменится ли значимость различий, если делать кластеризацию сразу по 2-м генам, по сравнению со значимостью кластеров отдельных генов? Выделим две выборки, и по паре аллелей генов А и В и нажмем кнопку Выполнить Запрос, см.рис.23
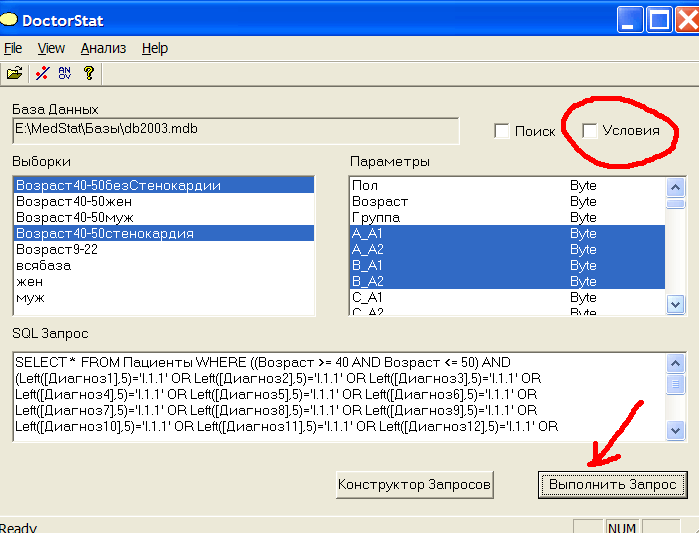
Рис.23 Анализ генотипов генов А и В.
В Excel появится более двухсот генотипов, часть из которых показана на рис.24:
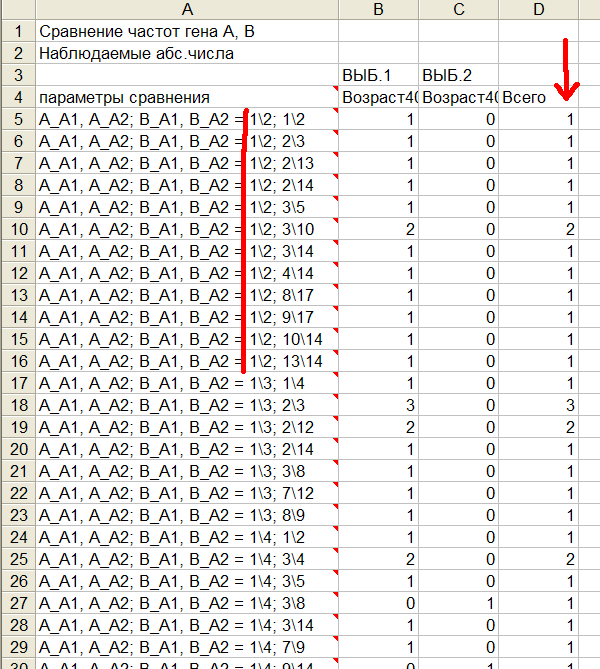
Рис.24 Генотипы двух генов.
Количество 2-х генных генотипов резко возросло по сравнению с количеством одногенных из-за сочетаний генов. Например, из рис.24 видно, что одному генотипу 1\2 гена А соответствует 12 различных генотипов В. Объем выборки остался прежним, а количество генотипов выросло, следовательно количество пациентов, приходящееся на один генотип, уменьшилось. Из рис.24 видно, что сумма пациентов (столбец D) с одинаковым генотипом в 2-х выборках не превосходит 3, а для большинства генотипов равна 1. В результате в таблице ожидаемых чисел почти все числа меньше 1. Малость большинства ожидаемых чисел делает невозможным кластеризацию, основанную на критерии хи-квадрат. При нажатиии клавиши Макрос, появляется окно с предупреждением, см.рис.25
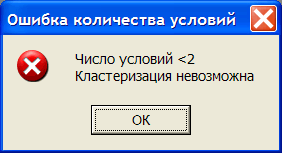
Рис.25 Ошибка кластеризации. Уменьшите мин. сумму B + C.
По умолчанию минимальная сумма В+С=5, см.рис.15 (ячейка F32). Количество генотипов, удовлетворяющих этому условию оказалось меньше 2, поэтому нечего кластеризовать. Для проведения кластеризации нужно уменьшить минимальную сумму, но тогда условия применимости хи-квадрат нарушатся. Вывод: для многогенного анализа объем выборок должен быть больше, чем при анализе отдельных генов.4. Отклонение частот генотипов от равновесия Харди-Вайнберга
Рассмотрим распределение частот генотипов в выборке для одного локуса (гена). В равновесии частота генотипа с точностью до множителя равна произведению частот аллелей. Отклонение частоты какого-либо генотипа от равновесной может иметь клиническое значение.Запустим программу DoctorStat. Проанализируем всех пациентов базы, т.е. рассмотрим выборку всябаза для гена А. В левом окне выделяем выборку всябаза, в правом - 1-ый аллель гена А. Если у Вас нет выборки всябаза, то создайте ее с помощью Конструктора Запросов. В качестве условия нужно выбрать Пол=0 ИЛИ Пол=1 (женщины + мужчины). В меню выбираем Гены->Равновесие или нажимаем комбинацию клавиш Ctrl+E. см.рис.26
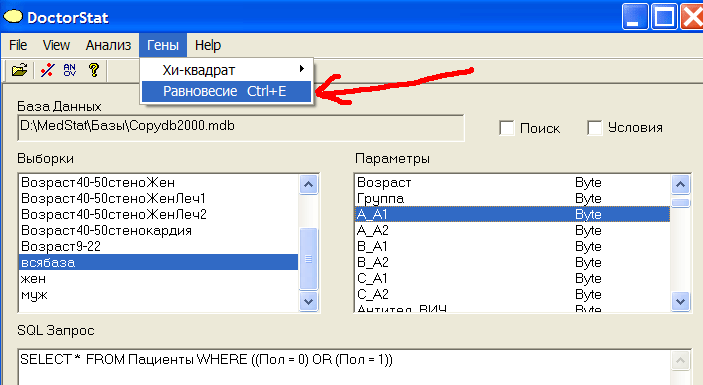
Рис.26 Анализ равновесия Харди_Вайнберга для гена А.
В Excel появится новый лист с гистограммой частот генотипов, см.рис.27
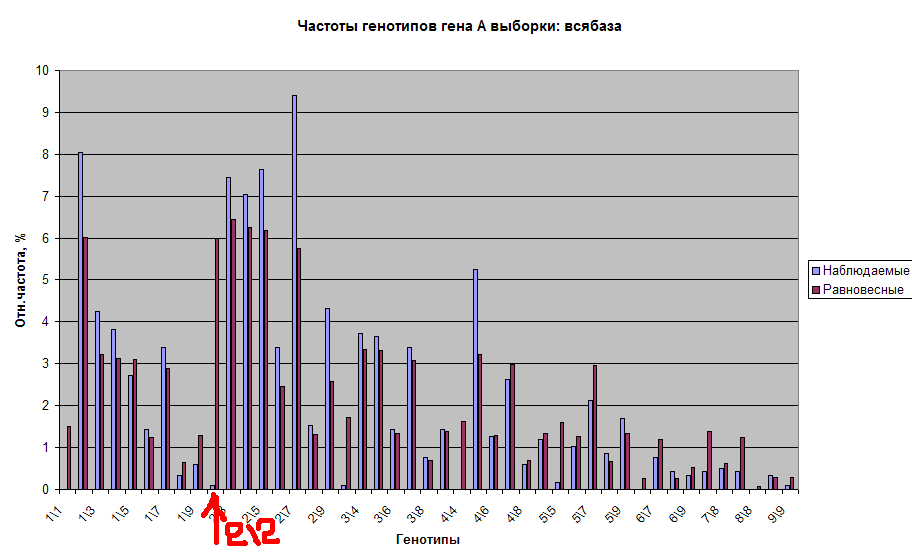
Рис.27 Гистограмма наблюдаемых и равновесных частот.
Обратите внимание на генотип 2\2, выделенный красной стрелкой. Равновесная частота (красный столбец) много больше наблюдаемой частоты (синий столбец). Нажмем кнопку Макрос. На Лист1 выведутся результаты анализа на равновесие Харди-Вайнберга, см.рис.28:
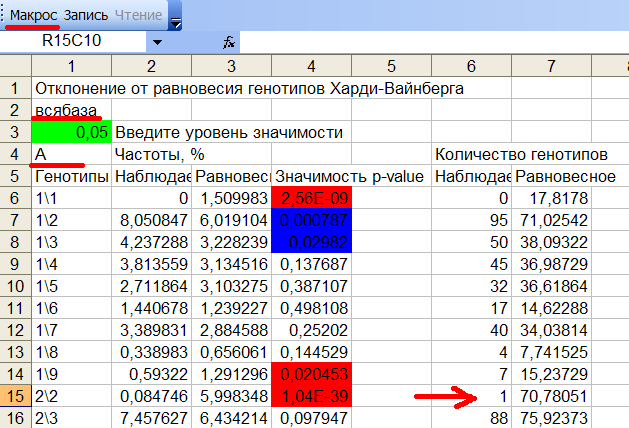
Рис.28 Верхняя часть Лист1.
Во 2-ой строке рис.28 показано название выборки всябаза. В 4-ой строке 1-ого столбца - название гена: А. В 3-ей строке (зеленая ячейка) можно менять (с последующим нажатием кнопки Макрос) пороговый уровень значимости p-value, ниже которого различия в индивидуальных частотах будут считаться достоверными и помечаться цветом. По умолчанию он равен 0,05. Во 2-ом столбце выведена наблюдаемая частота генотипа в %. В 3-ем столбце - ожидаемая (равновесная частота) генотипа. В 4-ом столбце - вычисленный для данного генотипа уровень значимости. Для генотипа 2\2 уровень значимости равен 1,04Е−39, что много меньше 0,05. Этот генотип встречается у одного пациента (6-ой столбец), а в равновесии должен был наблюдаться у 71 пациентов (7-ой столбец). По-видимому, гомозигота 2\2 обладает пониженной жизнеспособностью, поэтому до зрелого возраста доживает 1/71 часть от всех новорожденных. Если полученный уровень значимости меньше порогового и ожидаемое (равновесное) абсолютное число пациентов с данным генотипом больше 1 (7-ой столбец), то генотип считается неравновесным и ячейка p-value (4-ый столбец) окрашивается. В красный цвет, когда наблюдаемая частота генотипов меньше ожидаемой (генотип 2\2), в синий цвет, когда наблюдаемая частота больше ожидаемой (генотип 1\3). В нижней части Лист1 в желтой ячейке показана суммарная значимость теста на равновесие по всем генотипам, см.рис.29:
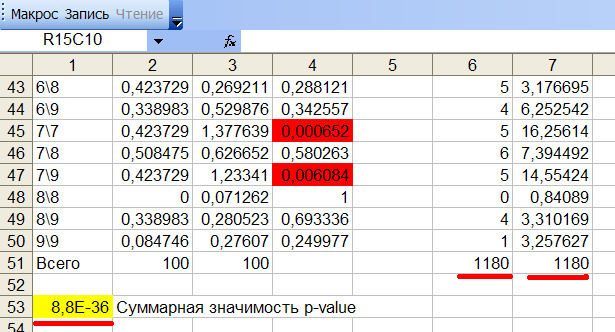
Рис.29 Нижняя часть Лист1.
В нашем случае суммарная значимость равна 8,8E-36. Это число много меньше порога 0,05. Это значит, что какие-то генотипы отклоняются от равновесия (они закрашены в красный и синий цвета в 4-ом столбце). Если суммарная значимость в желтой ячейке больше порога, то ВСЕ генотипы равновесны и рассматривать индивидуальные генотипы НЕ НУЖНО, даже если они выделены цветом как неравновесные. Внизу 6-ого и 7-ого столбца показан объем выборки всябаза = 1180 человек.